存力和算力一直以来是微电子和集成电路的核心业务。其中,随着大数据时代的到来,全球数据洪流对数据存储技术提出了严峻挑战。DNA具有极高的信息存储密度,每克DNA可存储数百艾字节的数据,并能在适当条件下保存千年,远超现有电子存储介质的能力,国际上由微软、西部数据等巨头已建立DNA数据存储联盟。然而,传统的DNA读取效率低下,往往需要数天时间,难以实现实时读取。纳米孔测序技术能够以单分子方式读取DNA的信息,通过记录电流变化实时解析碱基序列,显著缩短了读取时间,满足了快速数据存储的需求。纳米孔测序虽具有便携性和实时性,但其高插入-缺失(indel)错误率成为数据存储中的主要挑战。因此,如何通过编码和解码算法,抑制纳米孔测序中固有的高插入-缺失错误率,具有重要实际意义。
近日,南方科技大学深港微电子学院李毅课题组在DNA存储中的编解码领域取得新进展,在国际学术期刊Nature Communications上发表题为“Composite Hedges Nanopores codec system for rapid and portable DNA data readout with high INDEL-Correction”的研究论文。
基于上述背景,李毅课题组提出了一种面向纳米孔测序的新型编解码器Composite Hedges Nanopores (CHN)。该编解码器显著增强了系统的错误纠正能力,能够在高错误率环境下有效恢复数据。本方案使纳米孔测序可以更为便捷的适用于信息存储,还为在极端环境中实现便携、高效的数据读取提供了可能性。因此,本编解码器研究为迎接未来信息存储和数据安全领域带来了新的契机。
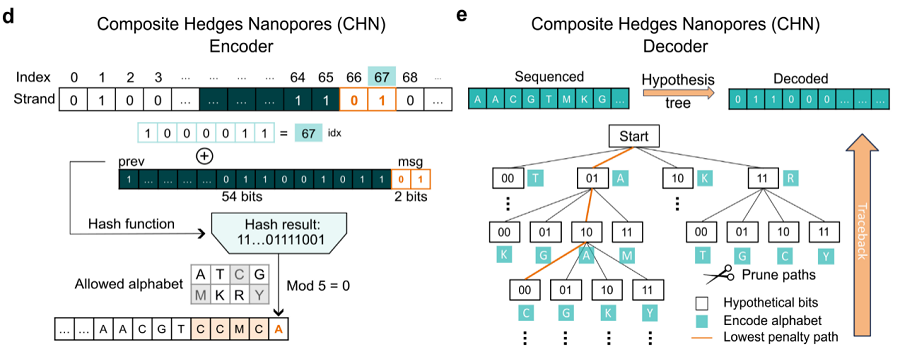
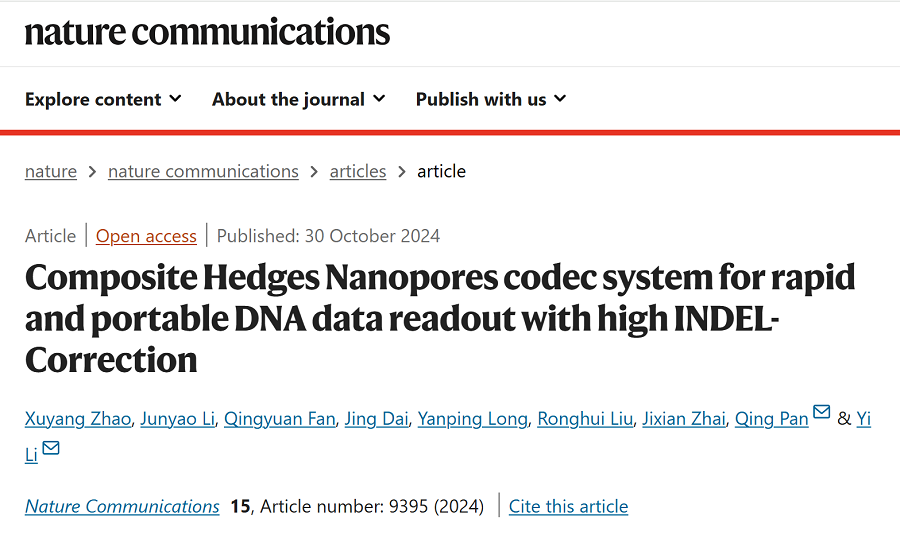
图 1 用于高插入/缺失错误校正的复合对冲纳米孔 (CHN) 编解码器架构。
图1展示了CHN编码方案的完整流程图及其在纳米孔编码系统中的性能表现。该编码流程通过使用简并碱基、嵌入锚点序列和约束筛选等方法,提升了DNA存储系统对插入-缺失错误的容忍度,有效降低了数据丢失风险。此外,通过与其他编码策略的比较,展现了CHN方案在容忍度和数据恢复率上的显著优势,表明其在高误差环境下的鲁棒性。
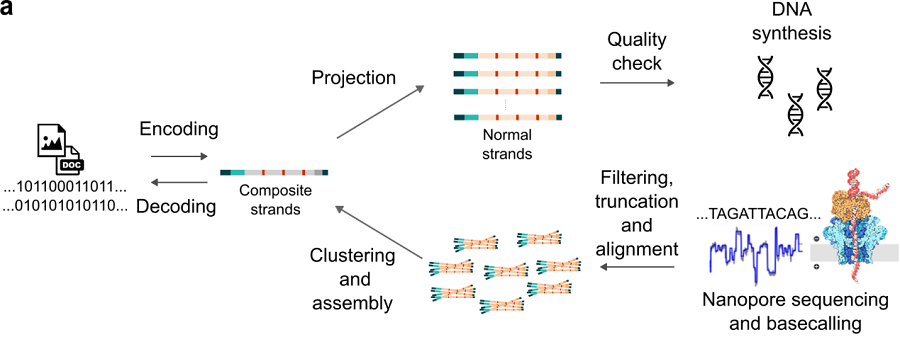
图 2 DNA数据恢复率与错误率模拟分析
图2深入探讨了在不同插入-缺失和替换错误率下的二进制数据恢复率。CHN编码系统在高达16%的插入-缺失率下仍能维持100%的数据恢复。
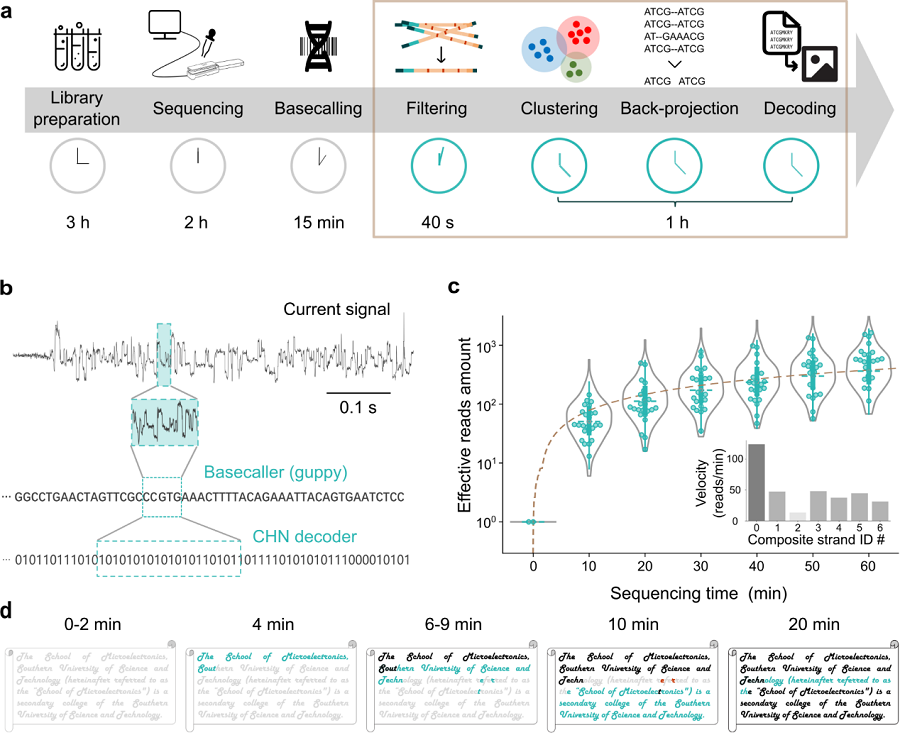
图 3 基于CHN编码的文本数据读取
图3呈现了基于CHN编码方案的体外文本数据恢复实验结果。实验显示,编码为7个复合链的文本文件在20分钟内实现了完整恢复。这一数据恢复时间的显著缩短,为加速DNA数据存储技术的实际应用提供了可能。此外,图3还通过纳米孔读数的有效分布,验证了CHN编码的高效性。

图 4 基于CHN编码的图像文件恢复及性能分析
图4展示了一个体外图像文件的CHN编码实验结果。该图像文件在120分钟内成功恢复,显示了CHN方案在更大数据文件上的强大恢复能力。该实验表明,尽管图像文件的恢复时间比文本文件稍长,但CHN系统依然在较低的覆盖度下表现出良好的数据完整性,为更大规模数据的DNA存储探索了新路径。
南方科技大学为论文第一单位,共同第一作者赵旭阳和李骏垚是本院2022级和2021级硕士研究生,目前均已完成学业并在课题组内攻读博士学位。南方科技大学李毅研究员、浙江工业大学潘清教授为论文共同通讯作者。本研究工作得到了国家重点研发计划生物与信息融合(“BT+IT”融合)专项青年科学家项目、国家自然科学基金、广东省联合基金、深圳市自然科学基金稳定支持项目等,以及浙江省和南方科技大学科学与工程计算中心的支持。/
文章来源:南方科技大学







