清华新闻网10月30日电 单分子荧光共振能量转移(FRET)技术是研究纳米尺度生物分子动态和相互作用的前沿生物物理工具。传统的长时间单分子FRET测量通常依赖于全内反射荧光(TIRF)显微镜,通过追踪单个荧光分子的强度变化,揭示分子层面的动态信息。然而,这一过程产生的大量数据对数据分析和筛选提出了挑战。传统的人工或半自动化方法不仅效率低下,而且容易引入主观偏差,严重依赖于实验者的经验。深度学习模型的应用则极大地减少了人为错误,提高了数据处理的速度和准确性。尽管如此,现有的深度学习模型往往难以根据用户的特定需求进行优化。
10月28日,清华大学生命科学学院和北京生物结构前沿研究中心陈春来课题组在《通讯·生物学》(Communications Biology)杂志发表了题为“基于深度学习和局部特征的单分子荧光事件自动识别方法”(Deep learning based local feature classification to automatically identify single molecule fluorescence events)的论文,介绍了一种名为DEBRIS(Deep lEarning Based fRagmentatIon approach for Single-molecule fluorescence event identification)的新方法,专注于对单分子荧光轨迹的局部特征进行分类。通过调整用户定义的标准,DEBRIS能够利用同一AI模型,精确筛选和分析四种不同类型的单分子荧光事件。
DEBRIS的核心理念在于,所有长时间尺度的单分子轨迹都是由有限类型的包含特定局部特征的短片段构建而成的。类似于俄罗斯方块中,有限的积木类型可以拼接出多种多样的复杂结构。DEBRIS模型中包含七个类别的双通道短片段强度-时间迹线,以模拟单分子双色荧光实验中常见的局部特征(图1a)。在应用时,DEBRIS采取滑动窗口逐帧预测局部特征,这一过程将强度-时间轨迹转换为模式-时间轨迹(图1b)。随后,根据用户定义的标准将其进一步分类为不同组别,并识别单分子事件。结合预测短片段的局部特征、滑动窗口技术和用户定义的标准,DEBRIS能够适应任意模式和任意长度的双通道荧光轨迹,为单分子荧光事件的自动识别提供了一种强大的工具。
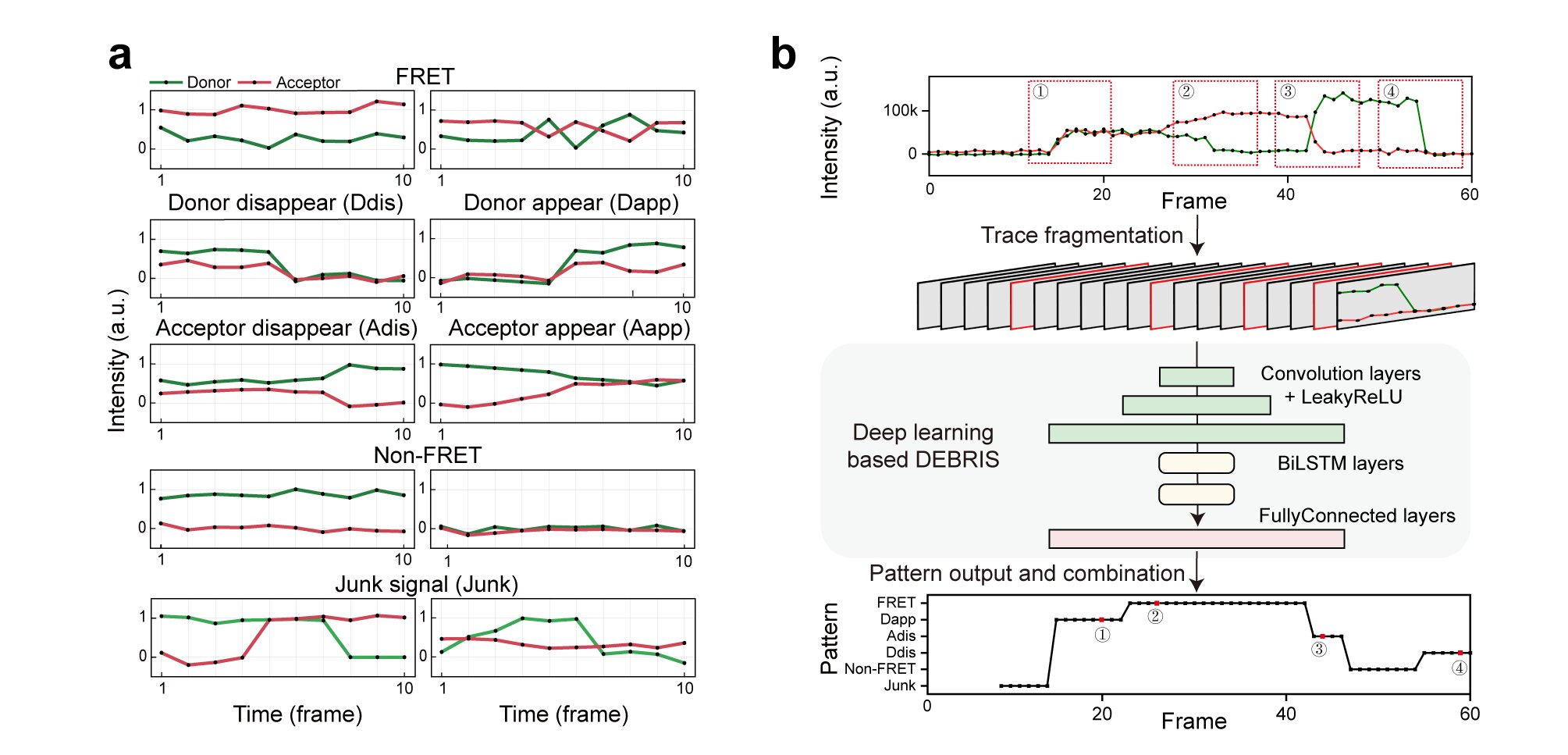
图1.DEBRIS的训练集构建和基本流程
为了评估DEBRIS在双色荧光轨迹上的分类性能,研究人员使用了五组单分子双色FRET数据集(图2)。以人工分类结果作为基准时,DEBRIS获得的FRET分布与人工分类结果相似性达到99%。这一结果凸显了DEBRIS的准确性,同时显示出DEBRIS在效率、客观性和可重复性方面具有显著优势。
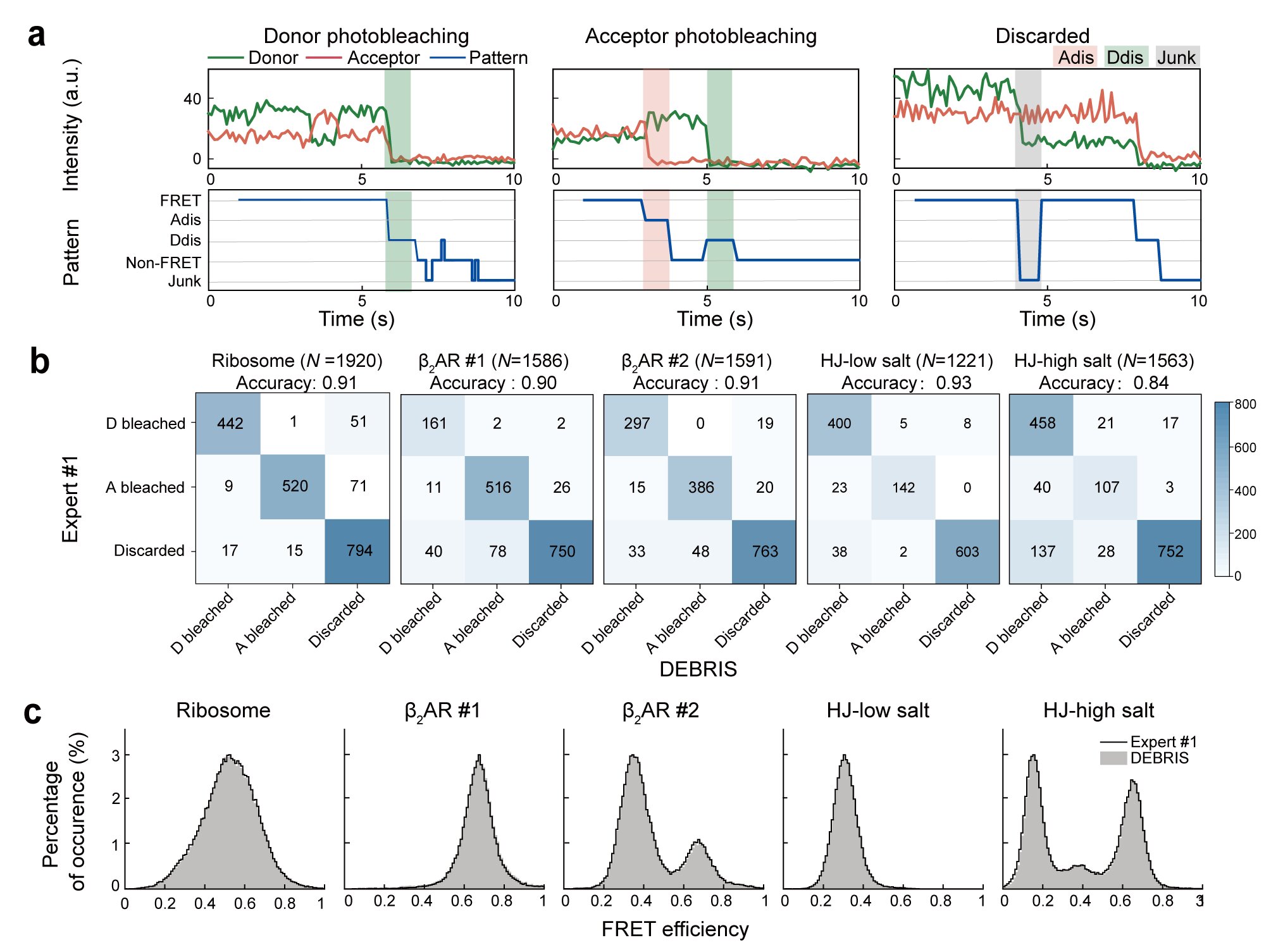
图2.DEBRIS分类性能评估
研究人员利用DEBRIS首次实现对动态出现的荧光信号的准确识别。动态单分子荧光信号是指在采集开始时并不存在荧光信号,但由于扩散的标记分子与表面固定的分子相互作用导致荧光信号在采集过程中出现(图3)。例如,在研究Cas12a对DNA的切割过程中,可以动态捕捉到Cas12a结合、切割和DNA片段解离等一系列动态过程(图3a)。但已发表的深度学习模型尚无法有效处理此类动态单分子荧光信号。研究人员展示了DEBRIS可自动识别出Cas12a的结合和切割事件,并基于分类结果生成了FRET概率随时间变化密度图,可表征切割过程中Cas12a的一系列复杂构象变化。
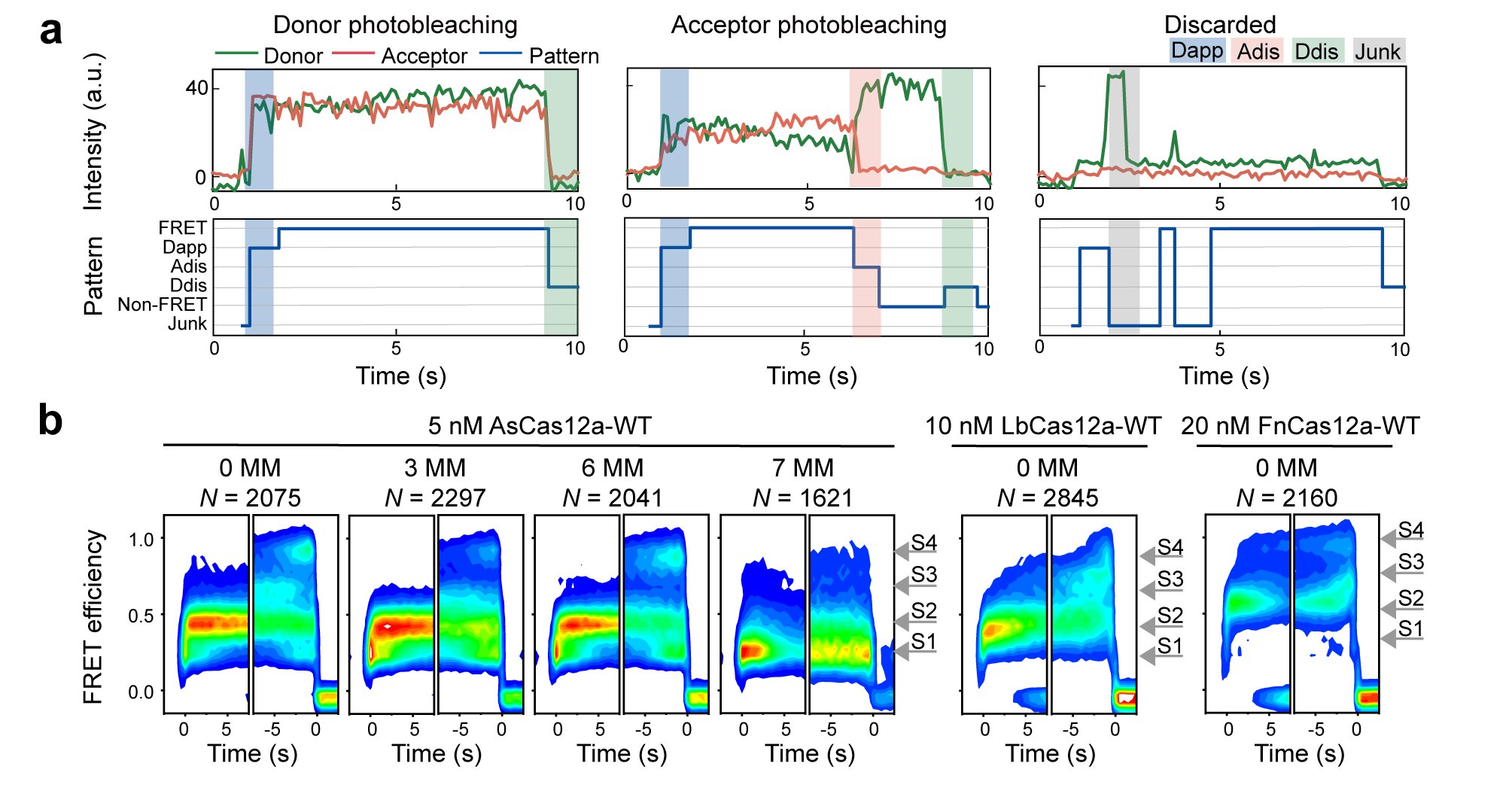
图3.DEBRIS在捕捉动态荧光信号中的应用
DEBRIS模型虽然是利用双色迹线进行训练的,但也可以拓展用于单色单分子荧光轨迹。研究人员对此也进行了验证(图4)
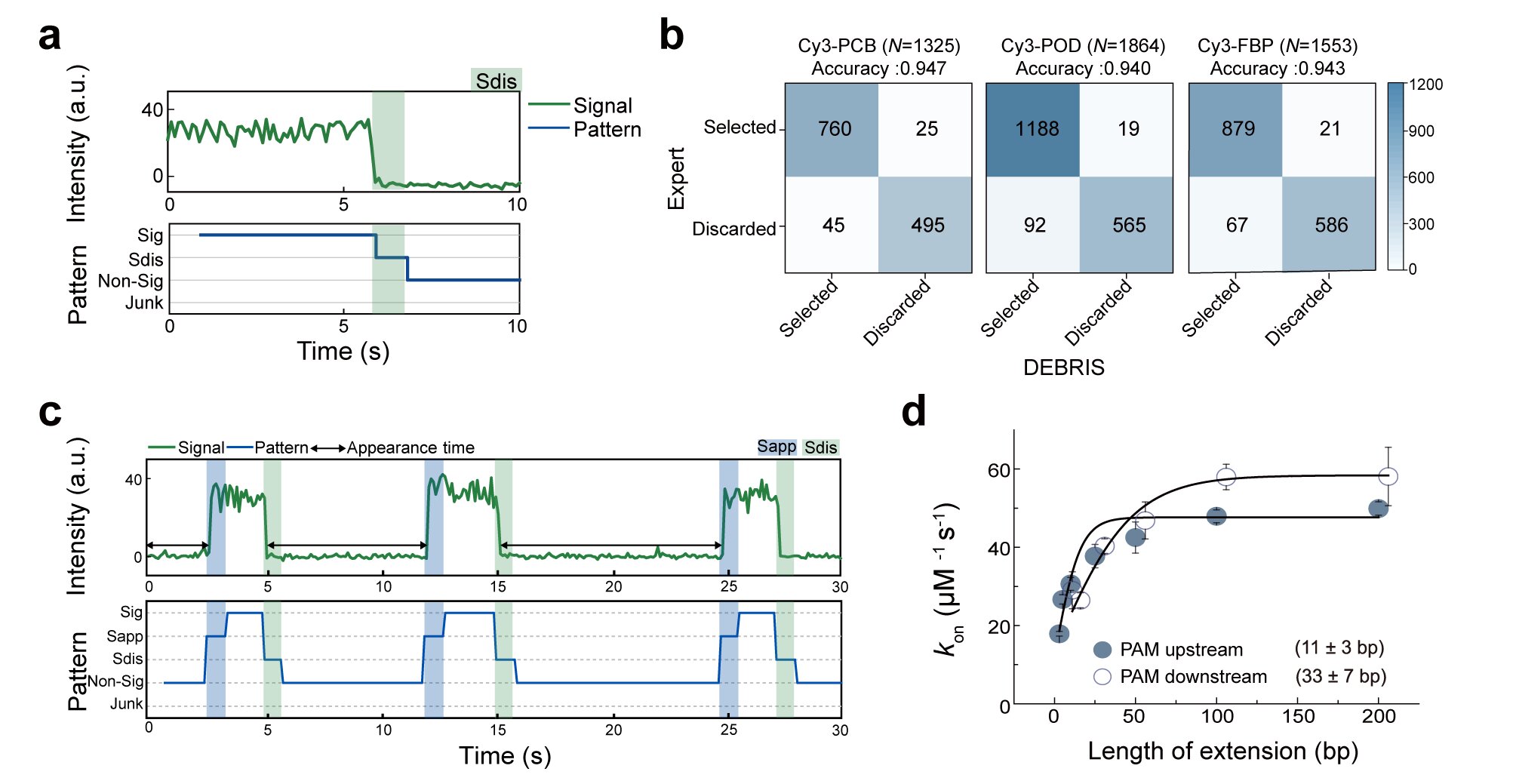
图4.DEBRIS在单色单分子荧光轨迹上的应用
DEBRIS通过精准识别单分子荧光轨迹的局部特征,并允许根据实验设计灵活调整分类标准,实现了在不修改神经网络结构的前提下,对双色/单色实验条件下的稳定和动态单分子荧光信号进行准确识别。这种创新的方法为单分子荧光技术的应用提供了一个高效的数据分析工具,极大地扩展了其在生物物理研究中的潜力。
清华大学生命科学学院陈春来副教授为论文通讯作者,生命科学学院2021级博士生周书棋为论文第一作者。生命科学学院2024届博士毕业生苗昱、2023级博士生邱浩仁和香港科技大学数学系教授姚远为研究提供了重要支持。研究得到国家自然科学基金委员会、国家自然科学基金委员会与香港研究资助局联合科研资助基金、北京生物结构前沿研究中心、膜生物学国家重点实验室的支持。
文章来源:清华大学









